利用全基因組關聯分析及系統生物學分析選拔生長初期耐寒基因發佈日期:2022/01/03
實施期間起迄
111年01月03日~111年12月30日執行單位
高崇峰副教授 / 國立中興大學農藝學系執行成果
大豆[Glycine max (L.) Merr.]富含脂質、蛋白質與多種人體所需之胺基酸,是目前全球重要的農藝作物之一。近年來,全球作物遭遇乾旱、寒害與淹水等非生物逆境,導致糧食生產供應風險增加。大豆是對低溫敏感的作物,國內大豆的種植分布受限於栽培期間氣候不確定性,因此,我們亟需一套新的育種策略,選育能因應氣候逆境且具高價值性狀之大豆品種。本研究使用材料選自於行政院農業委員會農業試驗所與中興大學農藝學系合作執行的「臺灣重要作物核心種原基因體資源開發及快速育種平臺之建置」計畫(以下簡稱大豆旗鑑計畫) (林等,2020)之研究成果,包括(1) 226個大豆蔬食特性之核心種原收集系(包含毛豆、台灣近年商業品系及其親本)建置、(2)大豆耐寒性田間試驗,試驗材料包括202個種原來自核心收集系、108個花青素近同源系、15個台灣近年商業品系及其親本、(3)大豆生長初期寒害性狀分級系統建置、以及(4)大豆核心種原180K Axiom® SoyaSNP array基因型資料建置。本研究以農業試驗所林大鈞博士提供之耐寒性田間試驗調查結果及其建立之耐寒嚴重等級資料為基礎,做為本研究大豆耐寒時序性狀資料建置之依據。同時,以國立中興大學農藝學系王強生教授建置之大豆基因型(180K Axiom® SoyaSNP array)資料,經由大豆樣本以及SNP基因型資料品質管制分析,建置GWAS最適分析平台,鏈結外表型及基因型資料,進行大豆耐寒時序性性狀之全基因體關聯定位分析(genome-wide association study, GWAS),選拔大豆耐寒候選基因。
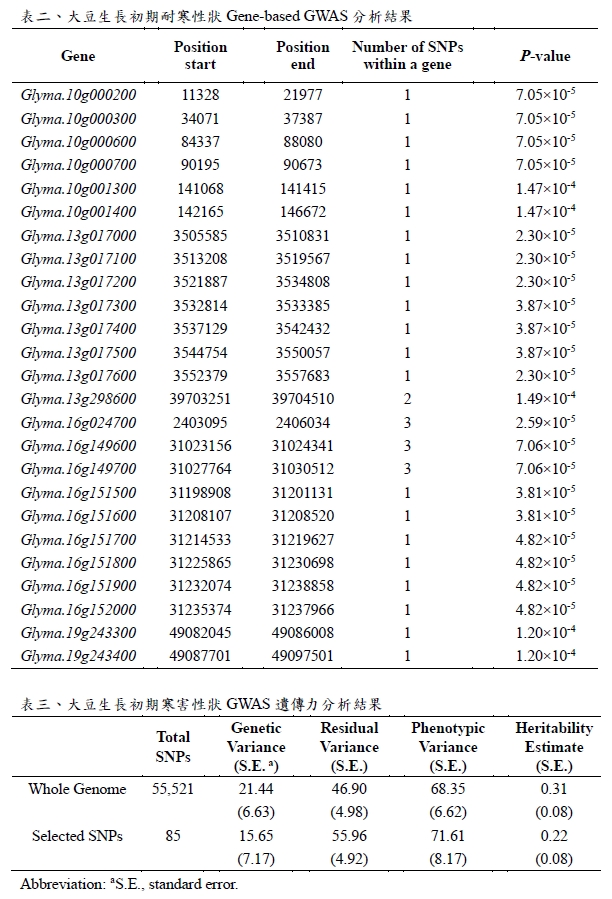
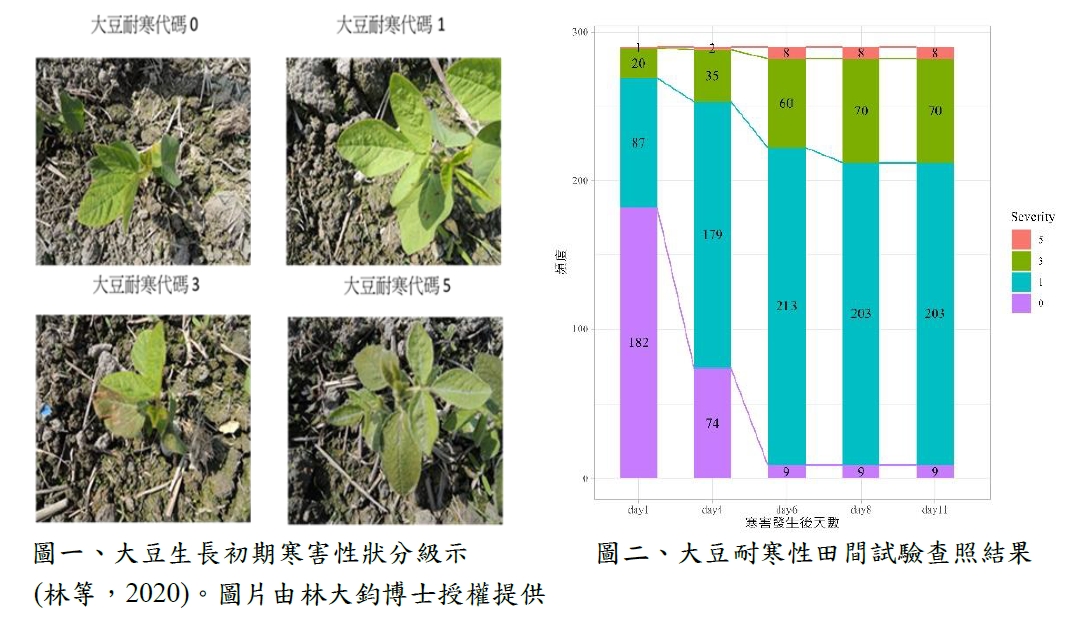
耐寒性田間試驗調查係觀察大豆植株在遭遇低溫寒害後(6.4℃),以目測方式觀察植株整體外表型變化,可區分為四個嚴重等級:0 (無影響)、1 (輕微影響)、3 (中等影響)、及5 (嚴重影響) (表一與圖一) (林等,2020)。根據調查結果(圖二),寒害性狀在寒害發生8天後各級分布變化幅度小,且耐寒性狀分數(圖三)亦與8天後各級分布趨於一致。
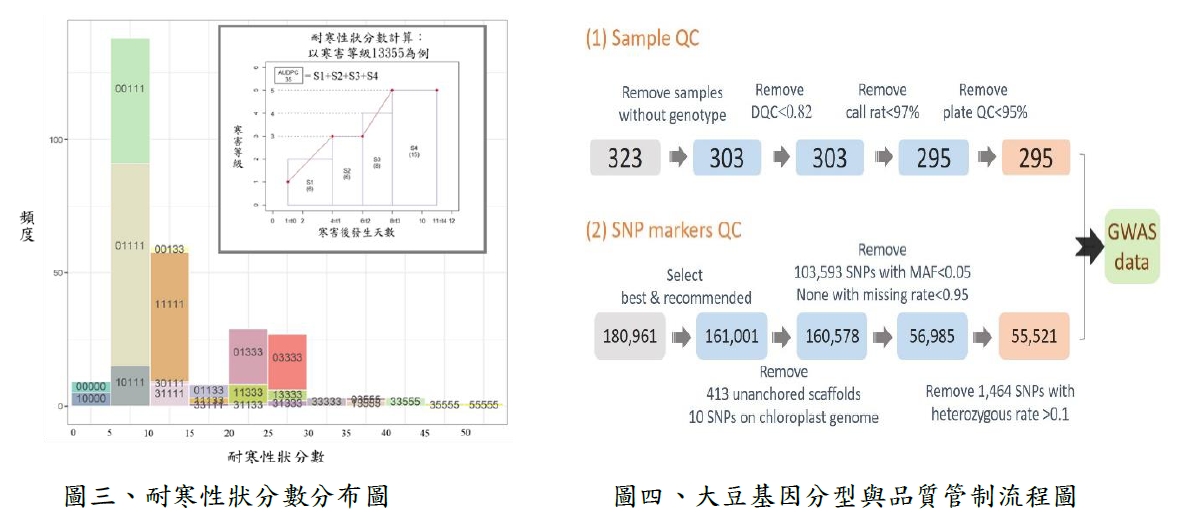
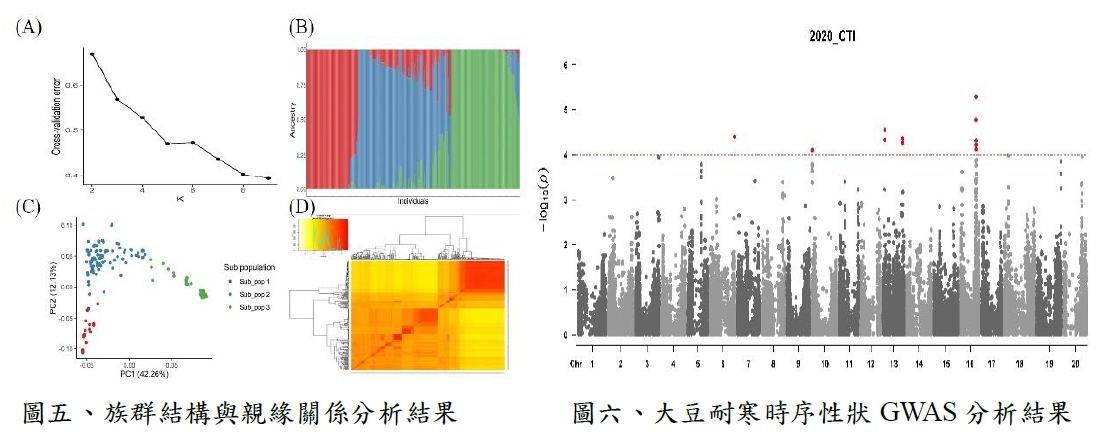
本研究使用SNP基因型資料經品質管制分析(圖四),共篩選出295個樣本與55,521個單核苷酸多態性(single nucleotide polymorphisms, SNPs)。根據族群結構分析(圖五A、B與C),設分群數K=9時之交叉驗證誤差為最小,表示分成9群的結果最佳。而主成分分析的結果顯示,第一主成分能夠解釋耐寒時序性狀42.26%的變異,第二主成分可以解釋12.13%的變異。在以K=3之結果結合主成分分析,可以得知2個主成分能夠將本研究之大豆族群明顯分為3個次族群。親緣關係熱圖也顯示大豆族群內部可分為3個次族群(圖五D)。因此,後續GWAS模型將納入族群結構與親緣關係訊息矩陣,做為GWAS模型校正項,降低GWAS分析結果偽陽性機會。
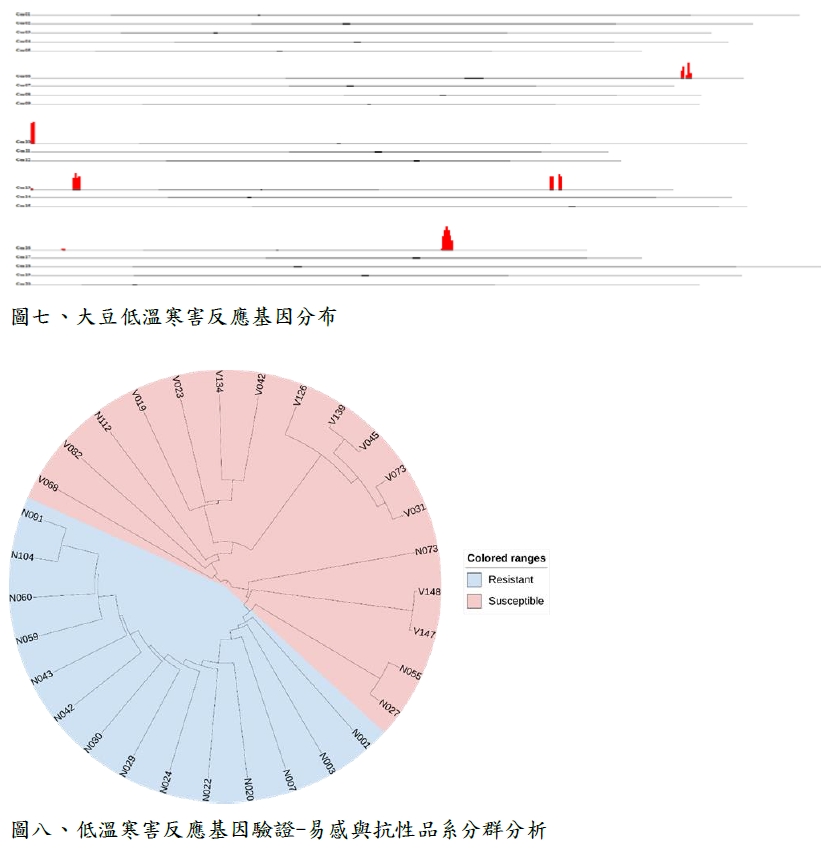
本研究利用線性混合模型分析大豆基因型(SNPs與基因)資料與耐寒時序性狀。首先,在SNP-based GWAS關聯定位分析,共計選拔出27個SNPs位點(座落在6、10、13及16號染色體)顯著與低溫寒害反應相關(P-value < 1×10-4) (圖六),我們進一步以該位點上、下游20Kb範圍,進行gene boundaries定位,結果計有72個基因被定位出來。在gene-level GWAS關聯定位分析(表二),計有25個基因(座落在第10、13、16及19號染色體)與低溫寒害反應顯著相關。整併SNP-level及gene-level GWAS結果,總計有75個大豆低溫寒害反應基因 (共有85個SNPs座落在這些基因內)進行遺傳力分析(表三),結果顯示這些低溫寒害反應相關之SNP與基因(85個SNPs)能夠解釋約22%(約占全基因體SNPs解釋力的71%)大豆耐寒時序性狀。
本研究進一步利用多變數分群分析,評估我們選拔的大豆低溫寒害反應基因(85個SNPs),發現這些SNPs能夠將17個易感品系從14個抗性品系有效地區分出來(圖八)。在這些選拔之大豆低溫寒害反應基因中,有1個基因被報導在轉殖GmDREB1B之大豆原生質體(protoplast),基因表現量有顯著增加(P-value < 0.01;fold change > 2) (Kidokoro et al., 2015)。DREB1B為轉錄因子並且會受到低溫處理誘導,在阿拉伯芥耐寒研究中,亦被報導參與調控耐寒相關基因。另,比對在Maruyama et al. (2012)研究中,生長3週之大豆以4℃處理1天並檢測基因表現量之結果,分別有5個及2個基因表現量呈現顯著正調控(false discovery rate < 0.05;fold change > 2)及顯著負調控 (false discovery rate < 0.05;fold change < 0.5)。研究結果顯示利用GWAS關聯定位分析,能夠快速選拔大豆低溫寒害反應基因,提供後續育種之利用。
參考文獻:
林大鈞、王強生、賴建成、高崇峰、游舜期、林思妤。2020。蔬食大豆迷你核心種原之建立 (第4年/全程4年)。行政院農業委員會農業試驗所109年度科技計畫研究報告。計畫編號:109農科-7.9.1-農-C2。
Kidokoro, S., K. Watanabe, T. Ohori, T. Moriwaki, K. Maruyama, J. Mizoi, N. Myint Phyu Sin Htwe, Y. Fujita, S. Sekita, K. Shinozaki, and K. Yamaguchi-Shinozaki. 2015. Soybean DREB1/CBF-type transcription factors function in heat and drought as well as cold stress-responsive gene expression. Plant J. 81: 505-518.
Maruyama, K., D. Todaka, J. Mizoi, T. Yoshida, S. Kidokoro, S. Matsukura, H. Takasaki, T. Sakurai, Y. Y. Yamamoto, K. Yoshiwara, M. Kojima, H. Sakakibara, K. Shinozaki, and K. Yamaguchi-Shinozaki. 2011. Identification of cis-acting promoter elements in cold- and dehydration-induced transcriptional pathways in arabidopsis, rice, and soybean. DNA Res. 19: 37-49.